Siamo ora al punto di rivedere il nostro processo evolutivo in base alle considerazioni illustrate nell'articolo precedente. Non più singoli nucleotidi, quindi, ma terne di nucleotidi, dette Codoni. Non più colorazione a 16 milioni di colori sulla base di 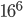 combinazioni delle 16 cifre esadecimali sulle 6 posizioni; ma, al suo posto, 20 colori dati dai 20 amminoacidi possibili (quindi con eventuale "sinonimia" di alcuni codoni rispetto alla codifica degli stessi amminoacidi).
combinazioni delle 16 cifre esadecimali sulle 6 posizioni; ma, al suo posto, 20 colori dati dai 20 amminoacidi possibili (quindi con eventuale "sinonimia" di alcuni codoni rispetto alla codifica degli stessi amminoacidi).
L'algoritmo stesso di determinazione casuale dell'evoluzione andrà integrato con la necessità di scartare i tre Codoni di Stop, che non possono essere ammessi all'interno delle sequenze di basi ammesse (e che i programmi stessi di analisi filogenetica considerano "non validi"). In altri termini l'algoritmo diventa:
- Vario casualmente (indicativamente con frequenza di 1 su 1.000) ogni base.
- Se nel codone di riferimento è effettivamente avvenuta una variazione, verifico che non sia diventato un Codone di Stop. In questo caso annullo la variazione (scelta evidentemente arbitraria);
- Sia che sia avvenuta una variazione ammessa, sia che non sia avvenuta nessuna variazione, rimappo (in funzione della sua rappresentazione) il codone secondo uno dei 20 colori corrispondenti ai 20 amminoacidi derivanti.
Prima di partire avremo quindi bisogno sia della tabella di conversione fra Codoni e Amminoacidi (e da qui, in modo ovviamente arbitrario, verso i 20 colori rappresentativi), sia dell'implementazione di un algoritmo di "derivazione" in un qualsiasi linguaggio di programmazione.
Partiamo con la tabella di correlazione. Occorre tenere presente che l'unica considerazione che ho tenuto a mente nello scegliere i colori è stata la loro resa (non troppo scuri, sufficientemente distinguibili l'uno dall'altro).
| Codone | Amminoacido | Codice HTML | Colore |
|---|---|---|---|
| TTT | Fenilalanina | #FFFFFF | |
| TTC | Fenilalanina | #FFFFFF | |
| TTA | Leucina | #E3F6CE | |
| TTG | Leucina | #E3F6CE | |
| CTT | Leucina | #E3F6CE | |
| CTC | Leucina | #E3F6CE | |
| CTA | Leucina | #E3F6CE | |
| CTG | Leucina | #E3F6CE | |
| ATT | Isoleucina | #A9F5BC | |
| ATC | Isoleucina | #A9F5BC | |
| ATA | Isoleucina | #A9F5BC | |
| ATG | Metionina | #FF0000 | |
| GTT | Valina | #F7BE81 | |
| GTC | Valina | #F7BE81 | |
| GTA | Valina | #F7BE81 | |
| GTG | Valina | #F7BE81 | |
| TCT | Serina | #FFFF00 | |
| TCC | Serina | #FFFF00 | |
| TCA | Serina | #FFFF00 | |
| TCG | Serina | #FFFF00 | |
| CCT | Prolina | #808000 | |
| CCC | Prolina | #808000 | |
| CCA | Prolina | #808000 | |
| CCG | Prolina | #808000 | |
| ACT | Treonina | #00FF00 | |
| ACC | Treonina | #00FF00 | |
| ACA | Treonina | #00FF00 | |
| ACG | Treonina | #00FF00 | |
| GCC | Alanina | #008000 | |
| GCA | Alanina | #008000 | |
| GCG | Alanina | #008000 | |
| GCT | Alanina | #008000 | |
| TAT | Tirosina | #00FFFF | |
| TAC | Tirosina | #00FFFF | |
| TAA | Codone di Stop (Ocra) | #808080 | |
| TAG | Codone di Stop (Ambra) | #808080 | |
| CAT | Istidina | #008080 | |
| CAC | Istidina | #008080 | |
| CAA | Glutammina | #8181F7 | |
| CAG | Glutammina | #8181F7 | |
| AAT | Asparagina | #2E64FE | |
| AAC | Asparagina | #2E64FE | |
| AAA | Lisina | #FF00FF | |
| AAG | Lisina | #FF00FF | |
| GAT | Acido aspartico | #800080 | |
| GAC | Acido aspartico | #800080 | |
| GAA | Acido glutammico | #FFA500 | |
| GAG | Acido glutammico | #FFA500 | |
| TGT | Cisteina | #FF4500 | |
| TGC | Cisteina | #FF4500 | |
| TGA | Codone di Stop (Opale) | #808080 | |
| TGG | Triptofano | #FFC0CB | |
| CGA | Arginina | #ADFF2F | |
| CGC | Arginina | #ADFF2F | |
| CGG | Arginina | #ADFF2F | |
| CGT | Arginina | #ADFF2F | |
| AGT | Serina | #FFFF00 | |
| AGC | Serina | #FFFF00 | |
| AGA | Arginina | #ADFF2F | |
| AGG | Arginina | #ADFF2F | |
| GGT | Glicina | #FA8072 | |
| GGC | Glicina | #FA8072 | |
| GGG | Glicina | #FA8072 | |
| GGA | Glicina | #FA8072 |
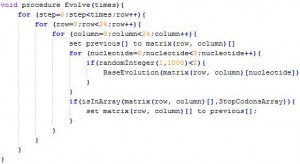
Quando allo pseudocodice da implementare, si tratta, ottimizzazioni a parte, di qualcosa come questo:
che a sua volta sfrutta la banale procedura di evoluzione puntuale:
In base alle impostazioni scelte (TTT che codifica per la Fenilalanina e Fenilalanina mappata sul bianco) abbiamo ancora un organismo iniziale completamente bianco, anche se di  quadratini.
quadratini.
Saranno però gli step successivi ad essere colorati diversamente: con meno colori, ma soprattutto con colori che compariranno più spesso (come quello di Arginina, Leucina e Serina, codificate da ben 6 codoni ognuna) e altri che compariranno con frequenza molto inferiore (come il Triptofano o la Metionina, codificati da un solo codone ognuna).
Nell'articolo seguente presenterò i risultati di questa nuova modalità di organizzare i dati e rappresentarli, traendo spunto dall'albero evolutivo che avevamo a suo tempo stabilito a tavolino.